
Intro
저는 개발 업무를 진행하면서 주로 Python을 사용해왔지만, 최근에 Rust를 접하게 되면서 Rust에 대해 궁금한 점이 생겼습니다.
Application 영역에서 Rust는 Python과 비교했을 때 어떤 차이가 있을까요?
Python은 간결한 문법과 강력한 라이브러리 생태계를 갖춘 언어로, 이미 여러 응용 분야에서 널리 사용되고 있습니다. 예를 들면 아래와 같은 분야들이 있습니다. 각 분야에 제공되는 library도 다양합니다.
- Web Development : Django, Flask, FastAPI
- Data Science : Numpy, Scikit-learn, Pandas, Matplotlib
- AI, ML : Tensorflow, Pytorch, Keras
- Image Processing, Computing Vision : OpenCV-python
- GUI Programming : PyQt, , PySide, Tkinter
반면, Rust는 Application 영역보다는 하드웨어 성능을 최대한 활용하면서, 기존 C/C++에서 발생할 수 있는 런타임 오류를 방지할 수 있는 규칙을 가진 언어입니다. Rust의 자세한 내용은 아래 링크에서 확인하실 수 있습니다.
[Rust Tutorial] 1 - Introduce
Intro최근 들어 저는 Rust를 시작했습니다. 개발경력이 C embedded를 시작으로 해서 최근은 Python Application에 몸담고 있는 와중에 Rust를 왜 시작하게 되었을까요? Rust는 저에게 꽤 이상적인(실용적인지
tyoon9781.tistory.com
이러한 Rust를 몇 번 사용해 보면서, 문득 궁금한 점이 생겼습니다. 현재(2024-12-02) Python과 Rust의 속도 차이는 얼마나 날까요? 물론 엄밀한 측정을 하기는 어렵지만, 실전에 좀 더 적합한 테스트를 진행해보고자 합니다.
Environment
docker-compose를 활용해 환경을 구성했습니다.
[Dockerfile]
# 베이스 이미지 선택
FROM python:3.12-slim
# 필수 패키지 설치
RUN apt-get update \
&& apt-get install -y curl build-essential \
&& apt-get clean \
&& rm -rf /var/lib/apt/lists/*
# Rust 설치 (rustup 사용)
RUN curl https://sh.rustup.rs -sSf | sh -s -- -y
ENV PATH="/root/.cargo/bin:${PATH}"
# 작업 디렉토리 설정
WORKDIR /code
# Python과 Rust 코드를 컨테이너에 복사
COPY python/ ./python/
COPY rust/ ./rust/
# 기본 명령어
CMD ["tail", "-f", "/dev/null"]
[docker-compose.yml]
services:
benchmark:
build:
context: .
dockerfile: Dockerfile
container_name: benchmark_container
working_dir: /code
volumes:
- ./python:/code/python
- ./rust:/code/rust
docker-compose up을 통해 container에 python과 rust를 정상적으로 설치했습니다.
docker-compose up


Host PC의 HW환경은 다음과 같습니다.
- CPU : 12th Gen Intel(R) Core(TM) i7-12700F
Python, Rust에게 시킬 작업은 아래와 같습니다.
- File Write job (float data)
- File Read job (float data)
- Calculate (Heavy Matrix)
이 3가지를 수행하는 code를 작성해 보겠습니다. 작성할 코드는 다음과 같습니다.
- only python : python에서 제공하는 library 만으로 작업
- numpy : python library인 numpy로 연산 최적화
- python multi processing : numpy + multi processing
- only rust : rust에서 제공하는 library만으로 작업
- ndarray : rust library인 ndarray를 사용하여 연산 최적화
- rust multi processing : ndarray + multi processing
그럼 코드를 살펴보도록 하겠습니다.
Code : Only Python vs numpy
5000 x 5000개의 random한 float data를 다루는 code입니다.
python만으로 동작하는 것과 numpy로 동작하는 것을 비교했습니다.
[main.py]
from modules import only_python as op
from modules import np_module as nm
import os
def main():
## ===================================
## only python vs numpy
## ===================================
ROW, COL = 5000, 5000
DUMMY_CSV_NAME = "dummy_raw.csv"
DUMMY_CSV_NUMPY_NAME = "dummy_numpy.csv"
## only python
op.write_data(DUMMY_CSV_NAME, ROW, COL)
data = op.read_data(DUMMY_CSV_NAME)
result1 = op.calculation_stddev(data)
## numpy
nm.write_data(DUMMY_CSV_NUMPY_NAME, ROW, COL)
data = nm.read_data(DUMMY_CSV_NAME) ## read same data
result2 = nm.calculation_stddev(data)
print(f"only python : {result1}")
print(f"numpy : {result2}")
os.remove(DUMMY_CSV_NAME) if os.path.exists(DUMMY_CSV_NAME) else None
os.remove(DUMMY_CSV_NUMPY_NAME) if os.path.exists(DUMMY_CSV_NUMPY_NAME) else None
print()
if __name__ == "__main__":
main()
[util.py]
import time
import functools
def timer(section):
def decorator(func):
@functools.wraps(func)
def wrapper(*args, **kwargs):
start_time = time.time()
result = func(*args, **kwargs)
print(f"[{section}] leadtime : {time.time() - start_time:.4f} seconds")
return result
return wrapper
return decorator
[modules/only_python.py]
from util import timer
import csv
import random
@timer("only python : write data")
def write_data(file_name: str, row: int, col: int):
data = [[random.random() for _ in range(col)] for _ in range(row)]
with open(file_name, 'w', newline='') as f:
writer = csv.writer(f)
writer.writerows(data)
@timer("only python : read data")
def read_data(file_name: str) -> list[list[float]]:
data = []
with open(file_name, 'r') as f:
reader = csv.reader(f)
for row in reader:
data.append([float(val) for val in row])
return data
@timer("only python : calculation stddev average")
def calculation_stddev_average(data2d: list[list[float]]) -> float:
means = [sum(row) / len(row) for row in data2d]
std_devs = [
(sum((x - mean) ** 2 for x in row) / len(row)) ** 0.5
for row, mean in zip(data2d, means)
]
return sum(std_devs) / len(std_devs)
[modules/np_module.py]
import numpy as np
from util import timer
@timer("numpy : write data")
def write_data(file_name: str, row: int, col: int):
np.savetxt(file_name, np.random.random((row, col)), delimiter=',')
@timer("numpy : read data")
def read_data(file_name: str) -> np.ndarray:
return np.loadtxt(file_name, delimiter=',')
@timer("numpy : calculation sttdev average")
def calculation_stddev_average(data2d: np.ndarray) -> float:
return np.mean(np.std(data2d, axis=1))
결과는 다음과 같이 나왔습니다. 엄밀한 측정 방법은 아니지만 반복 시행을 해도 비슷한 경향성을 가집니다.
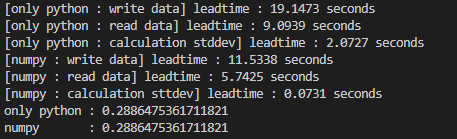
표로 비교하면 다음과 같습니다.
Numpy가 write, read로 2배정도빠르고, calculation은 비교가 어려울 정도로 차이가 심합니다.

이번에는 numpy와 multiprocessing을 비교해 보도록 하겠습니다. 얼핏 보기에는 당연히 multiprocessing이 빠르다고 생각되겠지만 실제로 얼마나 차이가 날지, 정말로 빠를지는 Code 동작을 확인 후 보도록 하겠습니다.
Code : numpy vs multiprocessing
2000 x 2000개의 random한 float data 8묶음을 다루는 code입니다.
numpy만 작성한 code vs multiprocess를 작성한 code를 비교해 보겠습니다.
[main.py]
from modules import multi_module as mm
from modules import np_module as nm
import os
def main():
## ===================================
## numpy list vs multi list
## ===================================
CPU_COUNT = 8
ROW, COL = 2000, 2000
DUMMY_CSV_NAME_LIST = [f"dummy_{i}.csv" for i in range(CPU_COUNT)]
DUMMY_CSV_NAME_MULTI_LIST = [f"dummy_{i}_multi.csv" for i in range(CPU_COUNT)]
## numpy list
nm.write_data_list(DUMMY_CSV_NAME_LIST, ROW, COL)
data_list = nm.read_data_list(DUMMY_CSV_NAME_LIST)
result1 = nm.calculation_inverse_matrix_list(data_list)
## multi list
mm.write_data_list(CPU_COUNT, DUMMY_CSV_NAME_MULTI_LIST, ROW, COL)
data_list = mm.read_data_list(CPU_COUNT, DUMMY_CSV_NAME_LIST) ## read same data
result2 = mm.calculation_inverse_matrix_list(CPU_COUNT, data_list)
## result
print(f"numpy list : {result1}")
print(f"multi list : {result2}")
for f1, f2 in zip(DUMMY_CSV_NAME_LIST, DUMMY_CSV_NAME_MULTI_LIST):
os.remove(f1) if os.path.exists(f1) else None
os.remove(f2) if os.path.exists(f2) else None
print()
print("Done")
if __name__ == "__main__":
main()
[modules/np.module.py]
from util import timer
import numpy as np
@timer("numpy list : write data")
def write_data_list(file_name_list: list[str], row: int, col: int):
[np.savetxt(file_name, np.random.random((row, col)), delimiter=',') for file_name in file_name_list]
@timer("numpy list : read data")
def read_data_list(file_name_list: list[str]) -> list[np.ndarray]:
return [np.loadtxt(file_name, delimiter=',') for file_name in file_name_list]
@timer("numpy list : calculation inverse matrix")
def calculation_inverse_matrix_list(data2d_list: list[np.ndarray]) -> float:
return np.mean([np.mean(np.linalg.inv(data2d)) for data2d in data2d_list])
[modules/multi_module.py]
import multiprocessing as mp
import numpy as np
from util import timer
def _worker_write_data(args):
file_name, row, col = args
np.savetxt(file_name, np.random.random((row, col)), delimiter=',')
@timer("multi list : write data")
def write_data_list(cpu_count: int, file_name_list: list[str], row: int, col: int):
with mp.Pool(processes=cpu_count) as pool:
pool.map(_worker_write_data, [(file_name, row, col) for file_name in file_name_list])
def _worker_read_data(file_name):
return np.loadtxt(file_name, delimiter=',')
@timer("multi list : read data")
def read_data_list(cpu_count: int, file_name_list: list[str]) -> list[np.ndarray]:
with mp.Pool(processes=cpu_count) as pool:
data_list = pool.map(_worker_read_data, file_name_list)
return data_list
def _worker_calculation_inverse_matrix(input_data):
return np.mean(np.linalg.inv(input_data))
@timer("multi list : calculation inverse matrix")
def calculation_inverse_matrix_list(cpu_count: int, data2d_list: list[np.ndarray]) -> float:
with mp.Pool(processes=cpu_count) as pool:
result_list = pool.map(_worker_calculation_inverse_matrix, data2d_list)
return np.mean(result_list)
결과는...? 충격젹인 결과입니다. multiprocess code가 더 느린 동작을 했습니다.

표로 비교하자면 다음과 같습니다.

왜 이렇게 되었을까요? numpy에서 제공하는 선형대수 연산은 사실 내부적으로 병렬적으로 돌아갑니다. 내부적으로 Test를 더 진행해본 결과, np.linalg.inv() 함수는 multiprocessing을 적용하지 않아도 core를 다 사용한다는 것을 확인할 수 있었습니다.

이렇게 동작하는 module을 multiprocessing으로 사용하게 되면 이미 full로 사용할 수 있는데 서로 자원을 뺏어가게 되는 상황이 벌어져 오히려 동작이 느려지는 상황을 확인할 수 있었습니다. 이렇게 자체적으로 python에서 multi thread활용을 하는 method에 대해서는 추가 자원을 할당할 때 조심히 사용해야 할 것 같습니다.
이런 calculation의 반전과는 다르게 read, write는 multiprocessing이 확실히 빠릅니다. 8core 할당에 3~4배 속도 향상은 확실히 눈이 띄는 향상폭입니다.
만약 단일 thread로 연산되는것이 확실하다면 multiprocessing으로 속도 향상을 기대해 볼 수 있습니다. 아래 코드를 추가해서 동작해보겠습니다.
[main.py]
...
## ===================================
## numpy list vs multi list (stddev)
## ===================================
CPU_COUNT = 8
ROW, COL = 2000, 2000
DUMMY_CSV_NAME_LIST = [f"dummy_{i}.csv" for i in range(CPU_COUNT)]
DUMMY_CSV_NAME_MULTI_LIST = [f"dummy_{i}_multi.csv" for i in range(CPU_COUNT)]
## numpy list
nm.write_data_list(DUMMY_CSV_NAME_LIST, ROW, COL)
data_list = nm.read_data_list(DUMMY_CSV_NAME_LIST)
result1 = op.calculation_stddev_list(data_list)
## multi list
mm.write_data_list(CPU_COUNT, DUMMY_CSV_NAME_MULTI_LIST, ROW, COL)
data_list = mm.read_data_list(CPU_COUNT, DUMMY_CSV_NAME_LIST) ## read same data
result2 = mm.calculation_stddev_list(CPU_COUNT, data_list)
## result
print(f"numpy list : {result1}")
print(f"multi list : {result2}")
for f1, f2 in zip(DUMMY_CSV_NAME_LIST, DUMMY_CSV_NAME_MULTI_LIST):
os.remove(f1) if os.path.exists(f1) else None
os.remove(f2) if os.path.exists(f2) else None
print()
...
[modules/only_python.py]
@timer("only python list : calculation stddev")
def calculation_stddev_list(data2d_list: list[list[float]]) -> float:
result_list = []
for data2d in data2d_list:
means = [sum(row) / len(row) for row in data2d]
std_devs = [
(sum((x - mean) ** 2 for x in row) / len(row)) ** 0.5
for row, mean in zip(data2d, means)
]
result_list.append(sum(std_devs)/len(std_devs))
return sum(result_list)/len(result_list)
[modules/multi_module.py]
def _worker_calculation_stddev(data2d: np.ndarray) -> float:
means = [sum(row) / len(row) for row in data2d]
std_devs = [
(sum((x - mean) ** 2 for x in row) / len(row)) ** 0.5
for row, mean in zip(data2d, means)
]
return sum(std_devs)/len(std_devs)
@timer("multi list : calculation stddev")
def calculation_stddev_list(cpu_count: int, data2d_list: list[np.ndarray]) -> float:
with mp.Pool(processes=cpu_count) as pool:
result_list = pool.map(_worker_calculation_stddev, data2d_list)
return sum(result_list)/len(result_list)
동작 결과 입니다. numpy 없이 연산을 진행하니 calculation에도 성능향상을 보였습니다.
(실효성이 있지는 않습니다. numpy가 월등하게 빠르기 때문에...)


다음 글에서는 rust로 똑같은 동작을 한 후 Python과 Rust를 비교해 보도록 하겠습니다. 감사합니다.
'Python > Story' 카테고리의 다른 글
| [Python] - numba가 빠르긴 빠릅니다 (0) | 2024.12.06 |
|---|---|
| Python - 파이썬의 선(The Zen of python) (0) | 2023.02.10 |
| 파이썬(Python)이란? (0) | 2023.02.08 |


