
Intro
Python은 쉽고 직관적인 문법 덕분에 많은 개발자들에게 사랑받는 Interpreter 기반의 언어입니다. 하지만, 순수 Python 코드의 실행속도는 C/C++, Go, Rust같은 Compile 기반의 언어에 비해 상당히 느립니다. 이런 느린 실행속도는 대규모 데이터를 처리할 때 매우 불리하게 작용합니다.
이러한 대규모 데이터를 빠르게 처리하기 위해서 Numpy를 사용하기도 합니다. Numpy는 C언어로 구현된 고성능 library로, Python만으로 구현된 코드와 비교하면 상당한 실행속도를 보입니다. 하지만 이는 Python의 자체 속도를 늘려주기 보다는 Numpy에서 제공하는 수학 공식과 관련된 함수들이 빠르게 동작하는 것에 의의가 있습니다. 본인이 작성한 알고리즘이 Numpy에서 지원하지 않는 알고리즘이라면 어떻게 해야 할까요? 여기서 Numba가 등장합니다.
Numba: A High Performance Python Compiler
Numba makes Python code fast Numba is an open source JIT compiler that translates a subset of Python and NumPy code into fast machine code. Learn More Try Numba »
numba.pydata.org
아래는 공식 홈페이지에서 가져온 Numba에 대한 설명입니다.
Numba is an open source JIT compiler that translates a subset of Python and NumPy code into fast machine code.
해석하자면 다음과 같습니다.
Numba는 Python 및 NumPy 코드의 하위 집합을 빠른 머신 코드로 변환하는 오픈 소스 JIT 컴파일러입니다.
이 문장을 제대로 이해하려면 우리는 아래 3가지의 개념에 대해서 명확히 알고 있어야 합니다.
- Numpy : C언어를 기반으로 만들어진 Python Library로, 수치 연산과 배열 처리에 최적화
- 머신 코드 : CPU에 직접 실행할 수 있는 Binary Code
- JIT(Just-in-time) Compile : 프로그램 실행시점(Runtime)에서 Compile을 수행하여 성능을 최적화 하는 기술.
JIT이 생소하신 분들을 위해 추가적인 설명
JIT(Just-In-Time) 컴파일러는 실행 시점에 컴파일을 수행하는 기술입니다. 일반적으로 컴파일러는 프로그램 실행 전에 소스 코드를 기계어로 변환하지만, JIT은 프로그램 실행 중 필요한 부분만을 실시간으로 컴파일하여 속도를 높입니다. 이 방식은 동적 언어(dynamic language)에서 특히 유용하며, 프로그램 실행 전에 최적화하기 어려운 코드를 실행 중에 최적화할 수 있습니다.
Python은 Interpreter 언어입니다. Interpreter는 코드를 한 줄씩 해석하고 실행하기 때문에, 전체 코드를 미리 Compile해서 생성된 Binary Code(머신 코드)보다 속도가 느릴 수밖에 없습니다.
(자세한 내용이 궁금하시면 아래 글 참조)
파이썬(Python)이란?
* 바쁘신 분들을 위한 3줄 요약 1. 파이썬은 매우 인기가 높은 프로그래밍 언어입니다. 2. 파이썬은 개발하기 쉬운 인터프리터 언어입니다. 3. 파이썬은 문법이 매우 직관적입니다. 소개 (Introduce)
tyoon9781.tistory.com
이런 상황에서는 다음과 같은 의문이 들 수 있습니다.
Python Code를 Compile하면 되는 것 아니냐?
실제로 이런 Idea를 기반으로 세상에는 여러 Python Code Compiler가 존재합니다. PyPy, Jython, IronPython, Cython등 Python Code를 Compile하는 기술들은 다양하게 존재합니다. 그러나 이런 Compiler를 사용하려면 몇 가지 문제가 발생할 수 있습니다.
- 호환성 문제 : Python은 기본적으로 Interpreter언어를 지향합니다. Compiler는 Python 표준이 나오면 그때서야 개발을 시작하기 때문에 뒤쳐질수밖에 없는 구조입니다. 실제로 Java기반 Python Compiler인 Jython은 3.x을 지원하지 않습니다
- 개발 편의성 저하 : Python은 쉽고 간결한 문법이 개발 속도 단축으로 이어지며 이는 곧 생산성이 높다는 것을 의미합니다. 하지만 Compiler 사용은 Minor한 개발 환경 구축, 추가적인 설정 등으로 생산성이 떨어질 수 있으며 이는 Python을 사용하면서 얻었던 장점들을 잃게 됩니다.
이런 문제들 때문에 Python의 본질을 훼손하지 않으면서도 실행 성능을 높이는 방법이 필요했습니다. 여기서 JIT Compile 기술을 활용하는 도구인 Numba가 등장합니다.
Code Example
numba를 사용하기 위해서는 pip로 numba를 설치해야 합니다. 이왕이면 numpy도 같이 설치합시다.
pip install numpy numba
코드는 다음과 같이 작성했습니다.
[main.py]
from modules import numba_module as nbm, numpy_module as npm, python_module as pym
def cal_2d_data():
raw_2d_data = npm.gen_2d_data(5000, 5000)
r0 = pym.calculation_stddev_2d(raw_2d_data)
r1 = pym.calculation_stddev_2d_list_comprehension(raw_2d_data)
r2 = npm.calculation_stddev_2d(raw_2d_data)
r3 = nbm.calculation_stddev_2d(raw_2d_data) ## compile time + runtime
r3 = nbm.calculation_stddev_2d(raw_2d_data) ## runtime
r4 = nbm.calculation_stddev_2d_nopython(raw_2d_data) ## compile time + runtime
r4 = nbm.calculation_stddev_2d_nopython(raw_2d_data) ## runtime
print(r0)
print(r1)
print(r2)
print(r3)
print(r4)
def cal_3d_data(): ## 100배 Data
raw_3d_data = npm.gen_3d_data(100, 5000, 5000)
# r0 = pym.calculation_stddev_3d(raw_3d_data) ## Too slow
r1 = npm.calculation_stddev_3d(raw_3d_data)
r2 = nbm.calculation_stddev_3d_parallel(raw_3d_data) ## compile time + runtime
r2 = nbm.calculation_stddev_3d_parallel(raw_3d_data) ## runtime
# print(r0)
print(r1)
print(r2)
def main():
cal_2d_data()
cal_3d_data()
if __name__ == "__main__":
main()
[util.py]
import time
import functools
def timer(section):
def decorator(func):
@functools.wraps(func)
def wrapper(*args, **kwargs):
start_time = time.time()
result = func(*args, **kwargs)
print(f"[{section}] lead time : {time.time() - start_time:.4f} seconds")
return result
return wrapper
return decorator
[modules/python_module.py]
from util import timer
import numpy as np
@timer("python : stddev 3d")
def calculation_stddev_3d(data_3d: np.ndarray) -> float:
## parallel keyword와 prange로 병렬처리를 진행한다.
data_len = len(data_3d)
result = 0.0
for n in range(data_len): ## prange로 data 병렬화
std_dev_sum = 0.0
num_rows = data_3d[n].shape[0]
num_cols = data_3d[n].shape[1]
for i in range(num_rows):
_mean = 0.0
for j in range(num_cols):
_mean += data_3d[n, i, j]
_mean /= num_cols
_square_sum = 0.0
for j in range(num_cols):
_square_sum += (data_3d[n, i, j] - _mean) ** 2
std_dev_sum += (_square_sum / num_cols) ** 0.5
result += std_dev_sum / num_rows
return result / data_len
@timer("python : stddev 2d")
def calculation_stddev_2d(data_2d: np.ndarray) -> float:
std_dev_sum = 0.0
num_rows = data_2d.shape[0]
num_cols = data_2d.shape[1]
for i in range(num_rows):
_mean = 0.0
for j in range(num_cols):
_mean += data_2d[i, j]
_mean /= num_cols
_square_sum = 0.0
for j in range(num_cols):
_square_sum += (data_2d[i, j] - _mean) ** 2
std_dev_sum += (_square_sum / num_cols) ** 0.5
return std_dev_sum / num_rows
@timer("python : stddev 2d list comprehension")
def calculation_stddev_2d_list_comprehension(data_2d: np.ndarray) -> float:
means = [sum(row) / len(row) for row in data_2d]
stddevs = [
(sum((x - mean) ** 2 for x in row) / len(row)) ** 0.5
for row, mean in zip(data_2d, means)
]
return sum(stddevs) / len(stddevs)
[modules/numpy_module.py]
import numpy as np
from util import timer
@timer("gen 2d data")
def gen_2d_data(row: int, col: int) -> np.ndarray:
return np.random.random((row, col))
@timer("gen 3d data")
def gen_3d_data(count: int, row: int, col: int) -> np.ndarray:
return np.random.random((count, row, col))
@timer("numpy : stddev 2d")
def calculation_stddev_2d(data_2d: np.ndarray) -> float:
return np.mean(np.std(data_2d, axis=1))
@timer("numpy : stddev 3d")
def calculation_stddev_3d(data_3d: np.ndarray) -> float:
return np.mean([np.std(data_2d, axis=1) for data_2d in data_3d])
[modules/numba_module.py]
import numpy as np
from numba import jit, njit, prange
from util import timer
@jit
def _jit_stddev_2d(data_2d: np.ndarray) -> float:
std_dev_sum = 0.0
num_rows = data_2d.shape[0]
num_cols = data_2d.shape[1]
for i in range(num_rows):
_mean = 0.0
for j in range(num_cols):
_mean += data_2d[i, j]
_mean /= num_cols
_square_sum = 0.0
for j in range(num_cols):
_square_sum += (data_2d[i, j] - _mean) ** 2
std_dev_sum += (_square_sum / num_cols) ** 0.5
return std_dev_sum / num_rows
@timer("numba : stddev 2d python")
def calculation_stddev_2d(data_2d: np.ndarray) -> float:
return _jit_stddev_2d(data_2d)
# @jit -> list comprehension 금지
# def _jit_stddev_2d_list_comprehension(data_2d: np.ndarray) -> float:
# means = [sum(row) / len(row) for row in data_2d]
# stddevs = [
# (sum((x - mean) ** 2 for x in row) / len(row)) ** 0.5
# for row, mean in zip(data_2d, means)
# ]
# return sum(stddevs) / len(stddevs)
# @timer("numba : stddev 2d list comprehension")
# def calculation_stddev_2d_list_comprehension(data_2d):
# return _jit_stddev_2d_list_comprehension(data_2d)
# @jit -> np.std는 지원하지 않는다.
# def _jit_stddev_2d_numpy(data_2d: np.ndarray) -> float:
# return np.mean(np.std(data_2d, axis=1))
# @timer("numba : stddev 2d numpy")
# def calculation_stddev_2d_numpy(data_2d):
# return _jit_stddev_2d_numpy(data_2d)
@njit
def _jit_stddev_2d_nopython(data_2d: np.ndarray) -> float:
## njit(=nopython mode)에서는 python 순수기능은 사용할 수 없다.
## list comprehension, python object(예 : list, dict, ...), f-string, try-except, ...
std_dev_sum = 0.0
num_rows = data_2d.shape[0]
num_cols = data_2d.shape[1]
for i in range(num_rows):
_mean = 0.0
for j in range(num_cols):
_mean += data_2d[i, j]
_mean /= num_cols
_square_sum = 0.0
for j in range(num_cols):
_square_sum += (data_2d[i, j] - _mean) ** 2
std_dev_sum += (_square_sum / num_cols) ** 0.5
return std_dev_sum / num_rows
@timer("numba : stddev 2d nopython")
def calculation_stddev_2d_nopython(data_2d: np.ndarray) -> float:
return _jit_stddev_2d_nopython(data_2d)
@njit(parallel=True)
def _jit_stddev_3d_parallel(data_3d: np.ndarray) -> float:
## parallel keyword와 prange로 병렬처리를 진행한다.
data_len = len(data_3d)
result = 0.0
for n in prange(data_len): ## prange로 data 병렬화
std_dev_sum = 0.0
num_rows = data_3d[n].shape[0]
num_cols = data_3d[n].shape[1]
for i in range(num_rows):
_mean = 0.0
for j in range(num_cols):
_mean += data_3d[n, i, j]
_mean /= num_cols
_square_sum = 0.0
for j in range(num_cols):
_square_sum += (data_3d[n, i, j] - _mean) ** 2
std_dev_sum += (_square_sum / num_cols) ** 0.5
result += std_dev_sum / num_rows
return result / data_len
@timer("numba : stddev 2d parallel")
def calculation_stddev_3d_parallel(data_3d):
return _jit_stddev_3d_parallel(data_3d)
이렇게 돌리면 다음과 같은 결과를 얻을 수 있습니다.
- 2D Data (5000 x 5000)
- 순수 python : 9.9513초
- list comprehension : 40.3798초
- numpy : 0.0899초
- numba : 0.6568초 (compile time + runtime)
- numba : 0.0246초 (runtime)
- numba + nopython : 0.1589초 (compile time + runtime)
- numba + nopython : 0.0364초 (runtime)
- 3D Data (100 x 5000 x 5000)
- numpy : 9.7122초
- numba : 1.5365초 (compile time + runtime)
- numba : 0.5619초 (runtime)
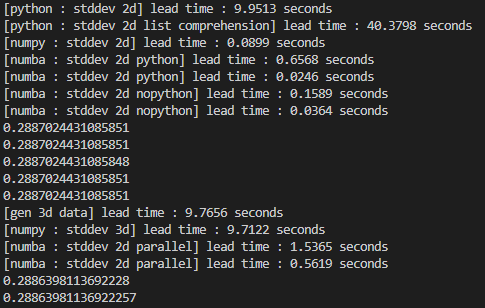
numba의 성능이 매우 빠름을 알 수 있습니다. 감사합니다.
* reference
'Python > Story' 카테고리의 다른 글
| [Python] only python vs numpy vs multiprocessing (0) | 2024.12.03 |
|---|---|
| Python - 파이썬의 선(The Zen of python) (0) | 2023.02.10 |
| 파이썬(Python)이란? (0) | 2023.02.08 |


